R语言绘制带ErrorBar的分组条形图代码的分享
目录
- 第一种实现方法:用aggregate计算数据
- 第二种实现方法:用dplyr包计算数据
笔者近期画了一张带error bar的分组条形图,将相关的代码分享一下。
感谢网友青山屋主的建议,提示笔者要严谨区分技术重复和生物学重复,所以笔者对文章做修改后重发。如果各位有任何建议,欢迎指正。
本文旨在给出一种利用R对生物学重复数据画带error bar的分组条形图的方法。
所用数据是模拟生成的:分成三个组,每个组进行了若干次生物学重复;测量的是3种基因的表达量。数据的部分内容如下:
## gene1 gene2 gene3 Group ## 1 49.72475 267.0007 126.2007 Group1 ## 2 114.62184 173.8780 150.2641 Group2 ## 3 128.03351 227.9456 152.6378 Group3 ## 4 134.90841 385.1979 148.2739 Group1 ## 5 136.56659 190.0663 122.6201 Group2 ## 6 143.88241 329.0516 236.9131 Group3
两种方法的完整代码放在了文末。如有问题,欢迎指正!
第一种实现方法:用aggregate计算数据
# 导入数据
setwd("E:/")
df <- read.csv("gene_exp.csv", header=T)
# 可以在这里改列名,这些列名就是最终图上X轴的标签名。
colnames(df)[1:3] <- c("gene-1", "gene-2", "gene-3")
str(df) # 显示数据集内容
## 'data.frame': 3000 obs. of 4 variables: ## $ gene-1: num 49.7 114.6 128 134.9 136.6 ... ## $ gene-2: num 267 174 228 385 190 ... ## $ gene-3: num 126 150 153 148 123 ... ## $ Group : Factor w/ 3 levels "Group1","Group2",..: 1 2 3 1 2 3 1 2 3 1 ...
# 将上述"宽数据"转化为"长数据"
library(reshape2)
df_reshape <- melt(df, id.vars=c("Group"))
str(df_reshape)
## 'data.frame': 9000 obs. of 3 variables: ## $ Group : Factor w/ 3 levels "Group1","Group2",..: 1 2 3 1 2 3 1 2 3 1 ... ## $ variable: Factor w/ 3 levels "gene-1","gene-2",..: 1 1 1 1 1 1 1 1 1 1 ... ## $ value : num 49.7 114.6 128 134.9 136.6 ...
# 获取三个组各个基因表达量的平均值
df_mean <- aggregate(df_reshape$value, list(Group=df_reshape$Group,
gene=df_reshape$variable), mean, na.rm=T)
# 获取三个组各个基因表达量的标准差
df_sd <- aggregate(df_reshape$value, list(Group=df_reshape$Group,
gene=df_reshape$variable), sd, na.rm=T)
# 合并mean和sd
colnames(df_mean)[3] <- "mean"
colnames(df_sd)[3] <- "sd"
df_stat <- merge(df_mean, df_sd, by=c("Group", "gene"))
str(df_stat)
## 'data.frame': 9 obs. of 4 variables: ## $ Group: Factor w/ 3 levels "Group1","Group2",..: 1 1 1 2 2 2 3 3 3 ## $ gene : Factor w/ 3 levels "gene-1","gene-2",..: 1 2 3 1 2 3 1 2 3 ## $ mean : num 120 249 149 119 250 ... ## $ sd : num 19.4 51.4 30.2 21.2 52.3 ...
# 画图
#直接在画图的语句中计算出error_bar所需的数据:
#(即下面的ymin=mean-sd和ymax=mean+sd语句)。
library(ggplot2)
dodge <- position_dodge(width=.9)
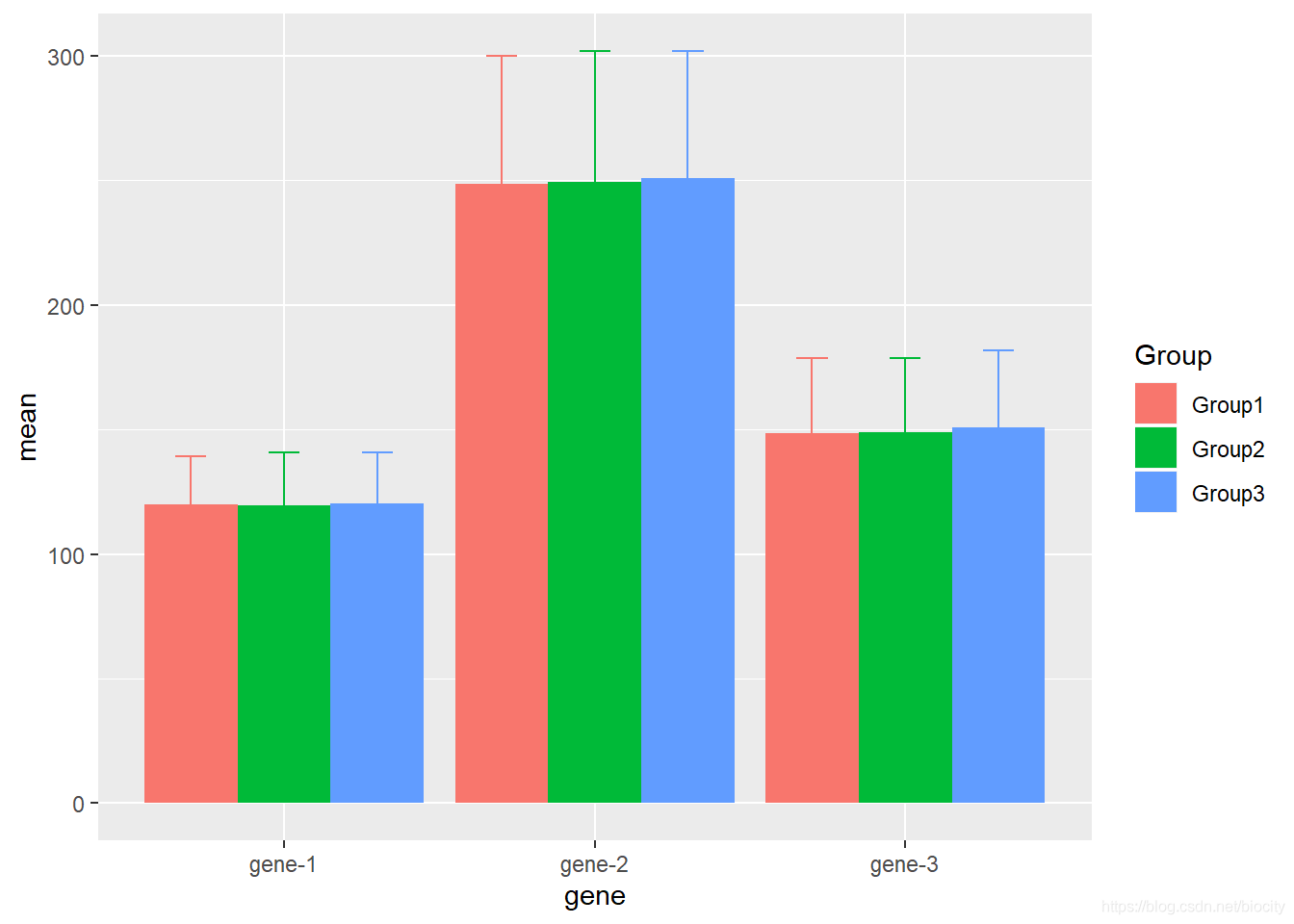
ggplot(data=df_stat) +
geom_bar(aes(x=gene, y=mean, fill=Group),
stat="identity", position=dodge) +
geom_errorbar(aes(x=gene, ymin=mean-sd, ymax=mean+sd, color=Group),
stat="identity", position=dodge, width=.3)
第二种实现方法:用dplyr包计算数据
# 导入数据
setwd("E:/")
df <- read.csv("gene_exp.csv", header=T)
# 可以在这里改列名,这些列名就是最终图上X轴的标签名。
colnames(df)[1:3] <- c("gene-1", "gene-2", "gene-3")
str(df) # 显示数据集内容
## 'data.frame': 3000 obs. of 4 variables: ## $ gene-1: num 49.7 114.6 128 134.9 136.6 ... ## $ gene-2: num 267 174 228 385 190 ... ## $ gene-3: num 126 150 153 148 123 ... ## $ Group : Factor w/ 3 levels "Group1","Group2",..: 1 2 3 1 2 3 1 2 3 1 ...
# 获取三个组各个基因表达量的平均值和标准差 library(tidyr) library(dplyr) df_stat <- tbl_df(df) %>% gather(gene, value, -Group) %>% # 将"宽数据"转化为"长数据" group_by(Group, gene) %>% # 将数据分组 summarise(mean=mean(value, na.rm=T), sd=sd(value, na.rm=T)) %>% # 计算每组数据的mean和sd ungroup() str(df_stat)
## Classes 'tbl_df', 'tbl' and 'data.frame': 9 obs. of 4 variables: ## $ Group: Factor w/ 3 levels "Group1","Group2",..: 1 1 1 2 2 2 3 3 3 ## $ gene : chr "gene-1" "gene-2" "gene-3" "gene-1" ... ## $ mean : num 120 249 149 119 250 ... ## $ sd : num 19.4 51.4 30.2 21.2 52.3 ...
# 画图
#直接在画图的语句中计算出error_bar所需的数据:
#(即下面的ymin=mean-sd和ymax=mean+sd语句)。
library(ggplot2)
dodge <- position_dodge(width=.9)
df_stat %>% ggplot() +
geom_bar(aes(x=gene, y=mean, fill=Group),
stat="identity", position=dodge) +
geom_errorbar(aes(x=gene, ymin=mean-sd, ymax=mean+sd, color=Group),
stat="identity", position=dodge, width=.3)
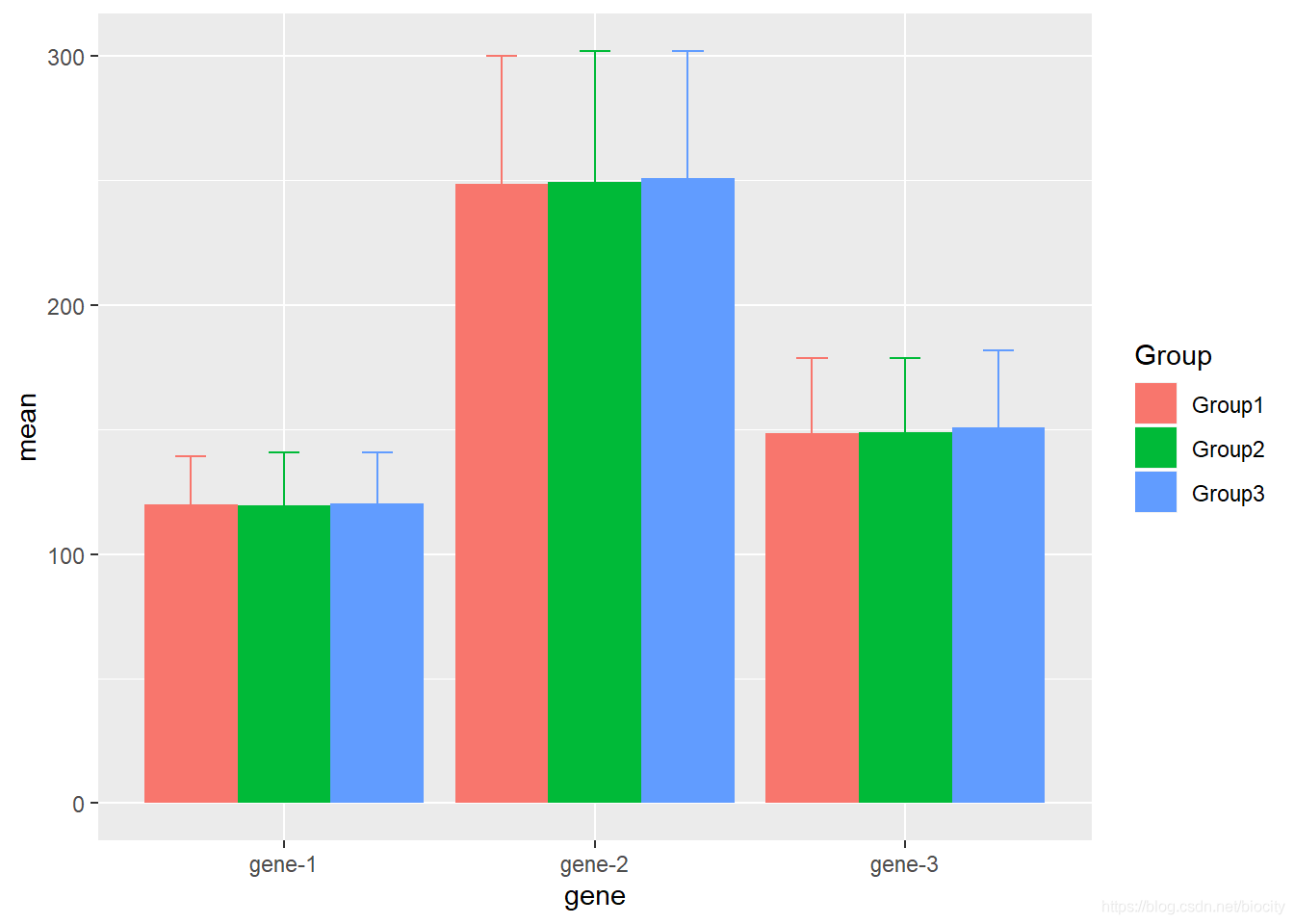
两种方法的结果是一样的,相对而言,dplyr的实现方法更简单快捷。
最后,两种方法的完整代码如下:
#################第一种实现方法:用aggregate计算数据######################
# 导入数据
setwd("E:/")
df <- read.csv("gene_exp.csv", header=T)
# 可以在这里改列名,这些列名就是最终图上X轴的标签名。
colnames(df)[1:3] <- c("gene-1", "gene-2", "gene-3")
str(df) # 显示数据集内容
# 将上述"宽数据"转化为"长数据"
library(reshape2)
df_reshape <- melt(df, id.vars=c("Group"))
str(df_reshape)
# 获取三个组各个基因表达量的平均值
df_mean <- aggregate(df_reshape$value, list(Group=df_reshape$Group,
gene=df_reshape$variable), mean, na.rm=T)
# 获取三个组各个基因表达量的标准差
df_sd <- aggregate(df_reshape$value, list(Group=df_reshape$Group,
gene=df_reshape$variable), sd, na.rm=T)
# 合并mean和sd
colnames(df_mean)[3] <- "mean"
colnames(df_sd)[3] <- "sd"
df_stat <- merge(df_mean, df_sd, by=c("Group", "gene"))
str(df_stat)
# 画图
#直接在画图的语句中计算出error_bar所需的数据:
#(即下面的ymin=mean-sd和ymax=mean+sd语句)。
library(ggplot2)
dodge <- position_dodge(width=.9)
ggplot(data=df_stat) +
geom_bar(aes(x=gene, y=mean, fill=Group),
stat="identity", position=dodge) +
geom_errorbar(aes(x=gene, ymin=mean-sd, ymax=mean+sd, color=Group),
stat="identity", position=dodge, width=.3)
####################第二种实现方法:用dplyr包计算数据######################
# 导入数据
setwd("E:/")
df <- read.csv("gene_exp.csv", header=T)
# 可以在这里改列名,这些列名就是最终图上X轴的标签名。
colnames(df)[1:3] <- c("gene-1", "gene-2", "gene-3")
str(df) # 显示数据集内容
# 获取三个组各个基因表达量的平均值和标准差
library(tidyr)
library(dplyr)
df_stat <- tbl_df(df) %>%
gather(gene, value, -Group) %>% # 将"宽数据"转化为"长数据"
group_by(Group, gene) %>% # 将数据分组
summarise(mean=mean(value, na.rm=T), sd=sd(value, na.rm=T)) %>% # 计算每组数据的mean和sd
ungroup()
str(df_stat)
# 画图
#直接在画图的语句中计算出error_bar所需的数据:
#(即下面的ymin=mean-sd和ymax=mean+sd语句)。
library(ggplot2)
dodge <- position_dodge(width=.9)
df_stat %>% ggplot() +
geom_bar(aes(x=gene, y=mean, fill=Group),
stat="identity", position=dodge) +
geom_errorbar(aes(x=gene, ymin=mean-sd, ymax=mean+sd, color=Group),
stat="identity", position=dodge, width=.3)
以上就是 R语言绘制带ErrorBar的分组条形图代码的分享的详细内容,更多关于 R语言绘制带ErrorBar的分组条形图的资料请关注我们其它相关文章!
赞 (0)

