python 读取.nii格式图像实例
我就废话不多说了,大家还是直接看代码吧~
# encoding=utf8
'''
查看和显示nii文件
'''
import matplotlib
matplotlib.use('TkAgg')
from matplotlib import pylab as plt
import nibabel as nib
from nibabel import nifti1
from nibabel.viewers import OrthoSlicer3D
example_filename = '../ADNI_nii/ADNI_002_S_0413_MR_MPR____N3__Scaled_2_Br_20081001114937668_S14782_I118675.nii'
img = nib.load(example_filename)
print (img)
print (img.header['db_name']) #输出头信息
width,height,queue=img.dataobj.shape
OrthoSlicer3D(img.dataobj).show()
num = 1
for i in range(0,queue,10):
img_arr = img.dataobj[:,:,i]
plt.subplot(5,4,num)
plt.imshow(img_arr,cmap='gray')
num +=1
plt.show()
3D显示结果:
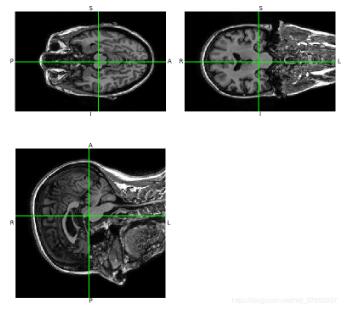
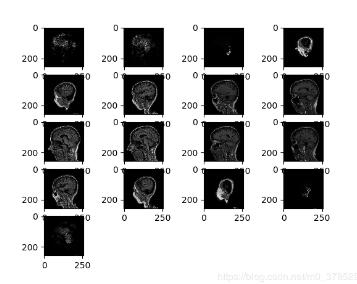
ADNI数据维度(256,256,170)分段显示:
补充知识:python nii图像扩充
我就废话不多说了,大家还是直接看代码吧~
import os
import nibabel as nib
import numpy as np
import math
src_us_folder = 'F:/src/ori'
src_seg_folder = 'G:/src/seg'
aug_us_folder = 'G:/aug/ori'
aug_seg_folder = 'G:/aug/seg'
img_n= 10
rotate_theta = np.array([0, math.pi/2])
# augmentation
aug_cnt = 0
for k in range(img_n):
src_us_file = os.path.join(src_us_folder, (str(k) + '.nii'))
src_seg_file = os.path.join(src_seg_folder, (str(k) + '_seg.nii'))
# load .nii files
src_us_vol = nib.load(src_us_file)
src_seg_vol = nib.load(src_seg_file)
# volume data
us_vol_data = src_us_vol.get_data()
us_vol_data = (np.array(us_vol_data)).astype('uint8')
seg_vol_data = src_seg_vol.get_data()
seg_vol_data = (np.array(seg_vol_data)).astype('uint8')
# get refer affine matrix
ref_affine = src_us_vol.affine
############### flip volume ###############
flip_us_vol = np.fliplr(us_vol_data)
flip_seg_vol = np.fliplr(seg_vol_data)
# construct new volumes
new_us_vol = nib.Nifti1Image(flip_us_vol, ref_affine)
new_seg_vol = nib.Nifti1Image(flip_seg_vol, ref_affine)
# save
aug_us_file = os.path.join(aug_us_folder, (str(aug_cnt) + '.nii'))
aug_seg_file = os.path.join(aug_seg_folder, (str(aug_cnt) + '_seg.nii'))
nib.save(new_us_vol, aug_us_file)
nib.save(new_seg_vol, aug_seg_file)
aug_cnt = aug_cnt + 1
############### rotate volume ###############
for t in range(len(rotate_theta)):
print 'rotating %d theta of %d volume...' % (t, k)
cos_gamma = np.cos(t)
sin_gamma = np.sin(t)
rot_affine = np.array([[1, 0, 0, 0],
[0, cos_gamma, -sin_gamma, 0],
[0, sin_gamma, cos_gamma, 0],
[0, 0, 0, 1]])
new_affine = rot_affine.dot(ref_affine)
# construct new volumes
new_us_vol = nib.Nifti1Image(us_vol_data, new_affine)
new_seg_vol = nib.Nifti1Image(seg_vol_data, new_affine)
# save
aug_us_file = os.path.join(aug_us_folder, (str(aug_cnt) + '.nii'))
aug_seg_file = os.path.join(aug_seg_folder, (str(aug_cnt) + '_seg.nii'))
nib.save(new_us_vol, aug_us_file)
nib.save(new_seg_vol, aug_seg_file)
aug_cnt = aug_cnt + 1
以上这篇python 读取.nii格式图像实例就是小编分享给大家的全部内容了,希望能给大家一个参考,也希望大家多多支持我们。
赞 (0)

